用python实现聚类分析
本文简单介绍如何用python里的库实现聚类分析
一键AI生成摘要,助你高效阅读
问答
·
一、简介
这里较为详细介绍了聚类分析的各种算法和评价指标,本文将简单介绍如何用python里的库实现它们。
二、k-means算法
和其它机器学习算法一样,实现聚类分析也可以调用sklearn中的接口。
from sklearn.cluster import KMeans
2.1 模型参数
KMeans(
# 聚类中心数量,默认为8
n_clusters=8,
*,
# 初始化方式,默认为k-means++,可选‘random’,随机选择初始点,即k-means
init='k-means++',
# k-means算法会随机运行n_init次,最终的结果将是最好的一个聚类结果,默认10
n_init=10,
# 算法运行的最大迭代次数,默认300
max_iter=300,
# 容忍的最小误差,当误差小于tol就会退出迭代(算法中会依赖数据本身),默认为0.0001
tol=0.0001,
# 是否将数据全部放入内存计算,可选{'auto', True, False},开启时速度更快但是更耗内存
# 'auto' : 当n_samples * n_clusters > 12million,不放入内存,否则放入内存,double精度下大概要多用100M的内存
precompute_distances='deprecated',
# 是否输出详细信息,默认为0
verbose=0,
# 用于随机产生中心的随机序列
random_state=None,
# 是否直接在原矩阵上进行计算
copy_x=True,
# 同时进行计算的核数(并发数),n_jobs用于并行计算每个n_init,如果设置为-1,使用所有CPU,若果设置为1,不并行,如果设置小于-1,使用CPU个数+1+n_jobs个CPU
n_jobs='deprecated',
# 可选的K-means距离计算算法, 可选{"auto", "full" or "elkan",default="auto"}
# full为欧式距离,elkan为使用三角不等式,效率更高,但不支持稀疏矩阵,当为稀疏矩阵时,auto使用full,否则使用elkan
algorithm='auto',
)
2.2 常用模型方法
fit(X)——对数据X进行聚类predict(X)——对新数据X进行类别的预测cluster_centers_——获取聚类中心labels_——获取训练数据所属的类别,比设置的聚类中心个数少1inertia_——获取每个点到聚类中心的距离和fit_predict(X)——先对X进行训练并预测X中每个实例的类,等于先调用fit(X)后调用predict(X),返回X的每个类transform(X)——将X进行转换,转换为K列的矩阵,其中每行为一个实例,每个实例包含K个数值(K为传入的类数量),第i列为这个实例到第K个聚类中心的距离fit_transform(X)——先进行fit之后进行transformscore(X)——输入样本(这里的样本不是训练样本,而是其他传入的测试样本)到他们的类中心距离和,然后取负数
2.3 实际例子
# 以two_moons数据为例
from sklearn.datasets import make_blobs
# 生成模拟的二维数据, X.shape——>(100, 2)
X, y = make_blobs(random_state=1)
# 设置为三个聚类中心
Kmeans = KMeans(n_clusters=3)
# 训练模型
Kmeans.fit(X)
2.3.1 获取聚类中心:
Kmeans.cluster_centers_
输出:
array([[ -1.4710815 , 4.33721882],
[ -6.58196786, -8.17239339],
[-10.04935243, -3.85954095]])
2.3.2 获取类别
Kmeans.labels_
输出:我们设置了3个聚类中心,所以输出3个类别。
array([0, 2, 2, 2, 1, 1, 1, 2, 0, 0, 2, 2, 1, 0, 1, 1, 1, 0, 2, 2, 1, 2,
1, 0, 2, 1, 1, 0, 0, 1, 0, 0, 1, 0, 2, 1, 2, 2, 2, 1, 1, 2, 0, 2,
2, 1, 0, 0, 0, 0, 2, 1, 1, 1, 0, 1, 2, 2, 0, 0, 2, 1, 1, 2, 2, 1,
0, 1, 0, 2, 2, 2, 1, 0, 0, 2, 1, 1, 0, 2, 0, 2, 2, 1, 0, 0, 0, 0,
2, 0, 1, 0, 0, 2, 2, 1, 1, 0, 1, 0], dtype=int32)
2.3.3 获取每个点到聚类中心的距离和
Kmeans.inertia_
输出:
156.28289251170003
三、mini batch k-means算法
mini batch k-means的用法和k-means类似。
from sklearn.cluster import MiniBatchKMeans
3.1 模型参数
MiniBatchKMeans(
n_clusters=8,
*,
init='k-means++',
max_iter=100,
# 每次采用数据集的大小
batch_size=100,
verbose=0,
# 计算训练样本的类
compute_labels=True,
random_state=None,
tol=0.0,
# 多少次迭代中质心没有变化,算法终止,默认10次
max_no_improvement=10,
# 用来候选质心的样本数据集大小,默认为batch_size的三倍
init_size=None,
# 用不同的初始化质心运行算法的次数。
# 这里和KMeans类意义稍有不同,KMeans类里的n_init是从相同训练集数据中随机初始化质心。
# 而MiniBatchKMeans类的n_init则是每次用不一样的采样数据集来跑不同的初始化质心运行。默认为3。
n_init=3,
# 某个类别质心被重新赋值的最大次数比例,为了控制算法的运行复杂度。分母为样本总数。如果取值较高的话算法收敛时间可能会增加,尤其是那些暂时拥有样本数较少的质心。默认是0.01。
reassignment_ratio=0.01,
)
四、层次聚类算法
同样使用sklearn接口
from sklearn.cluster import AgglomerativeClustering
4.1 模型参数
AgglomerativeClustering(
# 聚类中心的数量,默认为2
n_clusters=2,
*,
# 用于计算距离。可以为:’euclidean’,’l1’,’l2’,’mantattan’,’cosine’,’precomputed’,
# 如果linkage=’ward’,则affinity必须为’euclidean’
affinity='euclidean',
# 用于缓存输出的结果,默认为不缓存
memory=None,
connectivity=None,
# 通常当训练了n_clusters后,训练过程就会停止,但是如果compute_full_tree=True,则会继续训练从而生成一颗完整的树
compute_full_tree='auto',
# 计算两个簇之间的距离的方式,可选{'ward', 'complete', 'average', 'single'}
# 'ward':挑选两个簇来合并,使得所有簇中的方差增加最小
# 'complete':将簇中点之间最大距离最小的两个簇合并
# 'average':将簇中所有点之间平均距离最小的两个簇合并
# 'single':将簇中点之间最小距离最小的两个簇合并
linkage='ward',
# 链接距离阈值,在该阈值以上,簇将不会合并
# 如果不为None,那么n_clusters必须是None,而且compute_full_tree必须为True
distance_threshold=None,
# 计算簇之间的距离,可使树状图可视化
compute_distances=False,
)
4.2 模型常用方法
fit(X)——对数据X进行聚类labels_——获取训练数据所属的类别,比设置的聚类中心个数少1n_leaves_——层次树中的叶子数children_——一个大小为[n_samples-1,2]的数组,给出了每个非叶结点中的子节点数量fit_predict(X)——先对X进行训练并预测X中每个实例的类,等于先调用fit(X)后调用predict(X),返回X的每个类,该模型不能对新的数据点进行预测n_components_——一个整数,给出了连接图中的连通分量的估计
4.3 实际例子
from sklearn.cluster import AgglomerativeClustering
from sklearn.datasets import make_blobs
X, y = make_blobs(random_state=1)
agg = AgglomerativeClustering(n_clusters=3)
agg.fit_predict(X)
输出:对数据训练并预测
array([0, 2, 2, 2, 1, 1, 1, 2, 0, 0, 2, 2, 1, 0, 1, 1, 1, 0, 2, 2, 1, 2,
1, 0, 2, 1, 1, 0, 0, 1, 0, 0, 1, 0, 2, 1, 2, 2, 2, 1, 1, 2, 0, 2,
2, 1, 0, 0, 0, 0, 2, 1, 1, 1, 0, 1, 2, 2, 0, 0, 2, 1, 1, 2, 2, 1,
0, 1, 0, 2, 2, 2, 1, 0, 0, 2, 1, 1, 0, 2, 0, 2, 2, 1, 0, 0, 0, 0,
2, 0, 1, 0, 0, 2, 2, 1, 1, 0, 1, 0])
4.3.1 获取层次树中的叶子数
agg.n_leaves_
输出:
100
4.3.2 获取每个非叶结点中的子节点数量
agg.children_
输出:
array([[ 33, 68],
[ 35, 39],
[ 18, 21],
[ 30, 92],
[ 54, 58],
[ 49, 100],
[ 26, 55],
[ 23, 27],
[ 20, 45],
[ 3, 82],
[ 1, 71],
[ 16, 52],
[ 24, 38],
[ 22, 77],
[ 9, 59],
[ 44, 69],
[ 40, 106],
[ 15, 90],
[ 36, 94],
[ 53, 61],
[ 72, 108],
[ 37, 43],
[ 17, 78],
[ 60, 70],
[ 50, 102],
[ 76, 98],
[107, 114],
[ 56, 93],
[ 7, 110],
[ 63, 112],
[ 8, 66],
[ 11, 109],
[ 42, 84],
[ 5, 6],
[ 19, 121],
[ 97, 104],
[ 91, 105],
[ 79, 88],
[ 46, 73],
[113, 116],
[ 89, 132],
[ 85, 87],
[ 10, 41],
[ 29, 51],
[ 12, 96],
[ 34, 118],
[ 32, 144],
[115, 123],
[ 31, 48],
[ 62, 125],
[ 13, 130],
[ 81, 134],
[103, 135],
[ 0, 28],
[ 75, 142],
[120, 133],
[ 65, 117],
[ 47, 99],
[127, 137],
[101, 139],
[122, 136],
[ 74, 138],
[140, 148],
[ 80, 161],
[111, 119],
[155, 156],
[124, 129],
[131, 147],
[ 64, 151],
[141, 157],
[ 83, 95],
[143, 146],
[ 2, 168],
[ 67, 164],
[149, 170],
[ 86, 150],
[ 4, 14],
[128, 154],
[158, 167],
[ 25, 171],
[159, 165],
[160, 162],
[153, 169],
[ 57, 145],
[126, 152],
[163, 184],
[166, 177],
[173, 180],
[178, 183],
[175, 185],
[174, 187],
[176, 179],
[172, 188],
[181, 189],
[186, 192],
[190, 191],
[182, 193],
[194, 195],
[196, 197]])
4.3.3 可视化
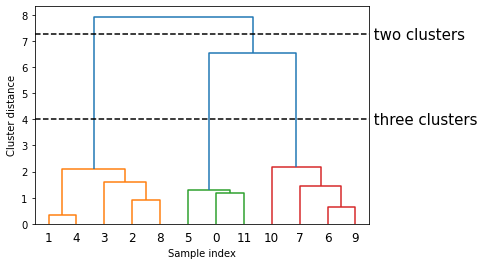
目前scikit-learn 没有绘制树状图的功能,但可以利用SciPy轻松生成树状图。SciPy的聚类算法接口与scikit-learn 的聚类算法稍有不同。SciPy提供了一个函数,接受数据数组X并计算出一个链接数组(linkage array),它对层次聚类的相似度进行编码。然后我们可以将这个链接数组提供给scipy 的dendrogram 函数来绘制树状图。
# 从SciPy中导入dendrogram函数和ward聚类函数
from scipy.cluster.hierarchy import dendrogram, ward
X, y = make_blobs(random_state=0, n_samples=12)
# 将ward聚类应用于数据数组X
# SciPy的ward函数返回一个数组,指定执行凝聚聚类时跨越的距离
linkage_array = ward(X)
# 现在为包含簇之间距离的linkage_array绘制树状图
dendrogram(linkage_array)
# 在树中标记划分成两个簇或三个簇的位置
ax = plt.gca()
bounds = ax.get_xbound()
ax.plot(bounds, [7.25, 7.25], '--', c='k')
ax.plot(bounds, [4, 4], '--', c='k')
ax.text(bounds[1], 7.25, ' two clusters', va='center', fontdict={'size': 15})
ax.text(bounds[1], 4, ' three clusters', va='center', fontdict={'size': 15})
plt.xlabel("Sample index")
plt.ylabel("Cluster distance")
输出:
五、DBSCN算法
from sklearn.cluster import DBSCAN
5.1 模型参数
DBSCAN(
# 数据点的邻域距离阈值(半径)
eps=0.5,
*,
# 数据点半径为eps的邻域中数据点个数的最小个数
min_samples=5,
# 可使用'euclidean', 'manhattan','chebyshev', 'minkowski”'
metric='euclidean',
metric_params=None,
# 最近邻搜索算法, 可选'auto', 'ball_tree', 'kd_tree', 'brute'
# 'brute'是使用蛮力搜索,一般使用'auto'即可,会自动拟合最好的最优算法
# 如果数据量很大或者特征也很多,用'auto'建树时间可能会很长,效率不高,建议选择KD树实现'kd_tree'
# 如果发现‘kd_tree’速度比较慢或者已经知道样本分布不是很均匀时,可以尝试用‘ball_tree’
# 如果输入样本是稀疏的,无论你选择哪个算法最后实际运行的都是‘brute’。
algorithm='auto',
# 使用KD树或者球树时, 停止建子树的叶子节点数量的阈值
# 这个值越小,则生成的KD树或者球树就越大,层数越深,建树时间越长,反之,则生成的KD树或者球树会小,层数较浅,建树时间较短
# 这个值一般只影响算法的运行速度和使用内存大小,因此一般情况下可以不管它。
leaf_size=30,
p=None,
n_jobs=None,
)
5.2 模型常用方法
fit(X)——对数据X进行聚类labels_——获取训练数据所属的类别,噪声点为-1fit_predict(X)——先对X进行训练并预测X中每个实例的类,等于先调用fit(X)后调用predict(X),返回X的每个类,该模型不能对新的数据点进行预测
六、聚类指标
6.1 RI 与 ARI
# RI 兰德指数
from sklearn.metrics.cluster import rand_score
# ARI 调整兰德指数
from sklearn.metrics.cluster import adjusted_rand_score
6.1.1 模型参数
rand_score(labels_true, labels_pred)
adjusted_rand_score(labels_true, labels_pred)
6.1.2 示例
rand_score([0, 0, 1, 1], [1, 1, 0, 0])
adjusted_rand_score([0, 0, 1, 1], [1, 1, 0, 0])
输出:
1.0
1.0
6.2 NMI
from sklearn.metrics.cluster import normalized_mutual_info_score
6.2.1 模型参数
normalized_mutual_info_score(
labels_true,
labels_pred,
*,
average_method='arithmetic',
)
6.2.2 示例
normalized_mutual_info_score([0, 0, 1, 1], [1, 1, 0, 0])
输出:
1.0
6.3 Jaccard系数
from sklearn.metrics import jaccard_score
6.3.1 模型参数
jaccard_score(
y_true,
y_pred,
*,
labels=None,
pos_label=1,
average='binary',
sample_weight=None,
zero_division='warn',
)
6.3.2 示例
jaccard_score([0, 0, 1, 1], [0, 0, 1, 1])
jaccard_score([0, 0, 1, 1], [1, 1, 0, 0])
输出:在使用前,需要转化为相同的类别标识
1.0
0.0
6.4 轮廓系数
from sklearn.metrics.cluster import silhouette_score
6.4.1 模型参数
silhouette_score(
X,
labels,
*,
metric='euclidean',
# 在数据的随机子集上计算轮廓系数时要使用的样本大小
sample_size=None,
random_state=None,
**kwds,
)
6.4.2 示例
silhouette_score(X, agg.labels_)
输出:agg.labels_为用AgglomerativeClustering算法对数据集X进行的聚类
0.5811444237627902
6.5 CH指标
from sklearn.metrics.cluster import calinski_harabasz_score
6.5.1 模型参数
calinski_harabasz_score(X, labels)
6.5.2 示例
calinski_harabasz_score(X, agg.labels_)
输出:
26.268277404270318
更多推荐
 已为社区贡献2条内容
已为社区贡献2条内容










所有评论(0)