Schrodinger Maestro分子对接详细教程 薛定谔软件 薛定谔 Maestro对接
chrodinger是药物发现的完整软件包,包括:基于受体和配体结构的诱导契合和柔性对接模式;基于受体结构及配体极性的对接模式;基于受体结构及溶液环境性质的对接模式;组合化学库设计及基于组合库的对接模式;基于配体结构的药物设计,药效团和3D-QSAR;生物分子结构模拟,蛋白、糖、核酸、小肽等;基于靶点的药物设计;ADME性质预测;
薛定谔分子对接教程
chrodinger是药物发现的完整软件包,包括:基于受体和配体结构的诱导契合和柔性对接模式;基于受体结构及配体极性的对接模式;基于受体结构及溶液环境性质的对接模式;组合化学库设计及基于组合库的对接模式;基于配体结构的药物设计,药效团和3D-QSAR;生物分子结构模拟,蛋白、糖、核酸、小肽等;基于靶点的药物设计;ADME性质预测;
参考:
Maestro 薛定谔软件使用:
- https://www.bilibili.com/video/BV1RN411X7Te
- https://www.youtube.com/watch?v=NkM8jjHr7f4&list=PL3dxdlKx_PcfuvHwJ0RjpZFt4HjwyTr7f

Maestro 薛定谔对接:
- https://www.bilibili.com/video/BV17p4y1h7mG
- https://space.bilibili.com/1119860191/channel/seriesdetail?sid=287811
额外注意:生成grid时候,如果有配体存在,按照氨基酸生成方式生成,记得删除掉配体,不然对接的时候会把配体当成蛋白一体的;如果按照氨基酸生成方式生成,没有配体,或者直接按照配体生成grid则都没影响
一、对接蛋白受体有小分子配体情况
1、下载pdb受体复合物,小分子也下载
这里下载的3ik3,里面的小分子也单独下载
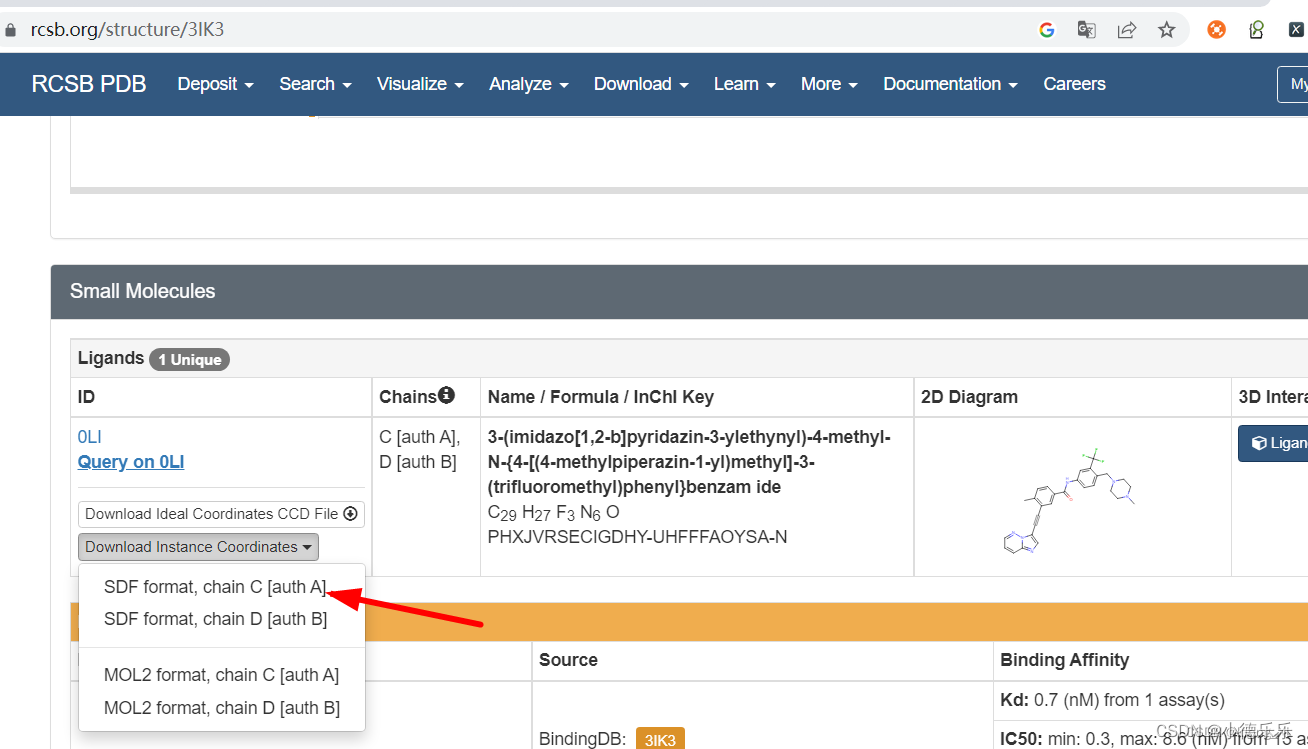
2、Maestro加载受体配体,首先配体小分子预处理,模块LigGprep,对小分子能量最小化,生成3D构象
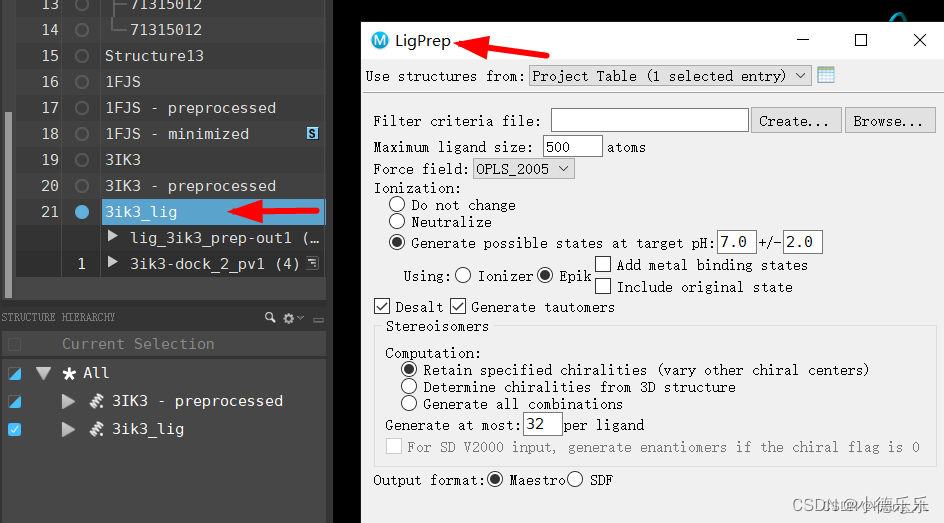
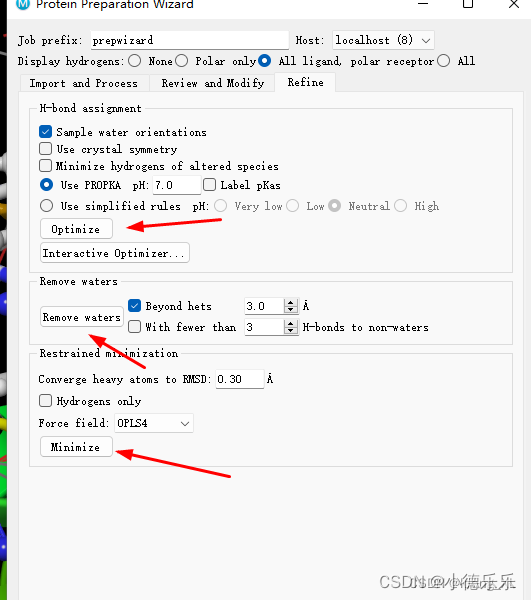
3、然后受体蛋白继续处理,模块Protein Preparation Wizard,结束后左侧会生成处理后结果
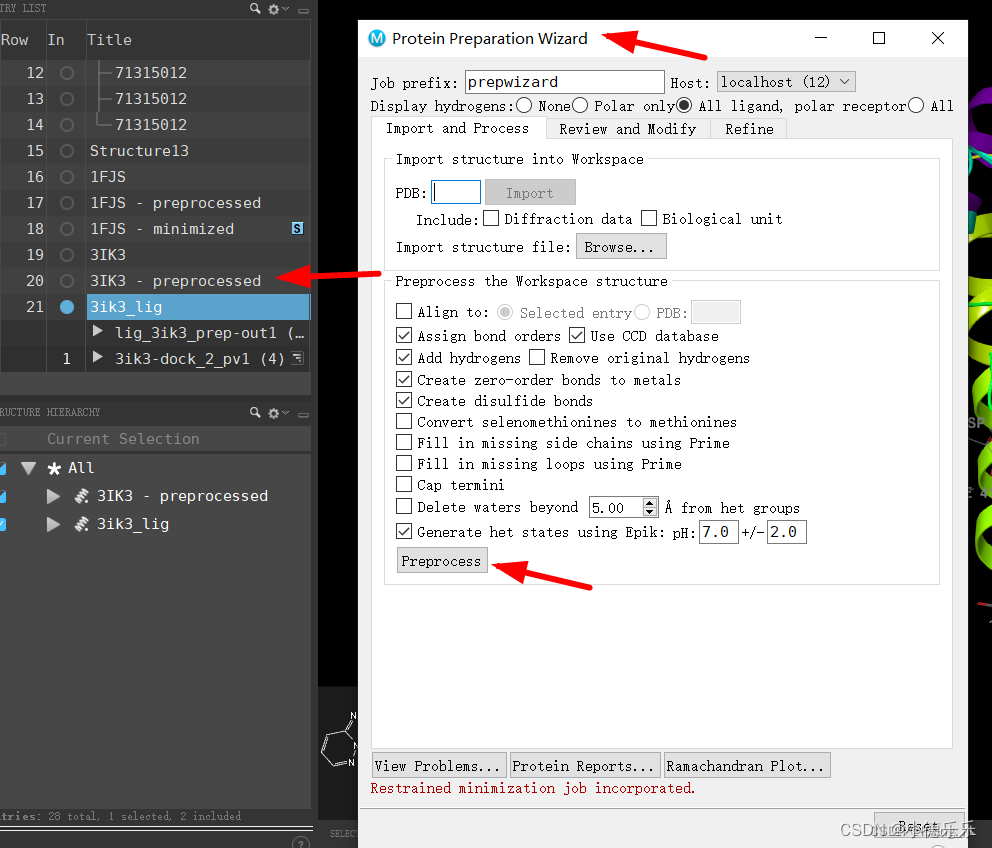
Refine这里也需要操作才能完整对蛋白预处理优化
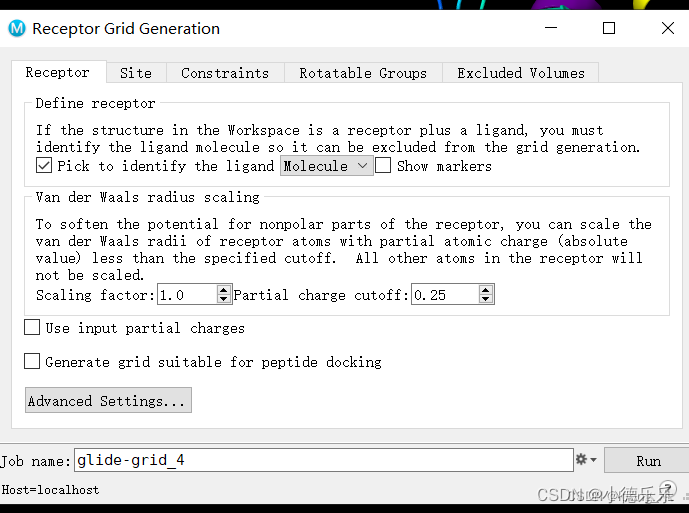
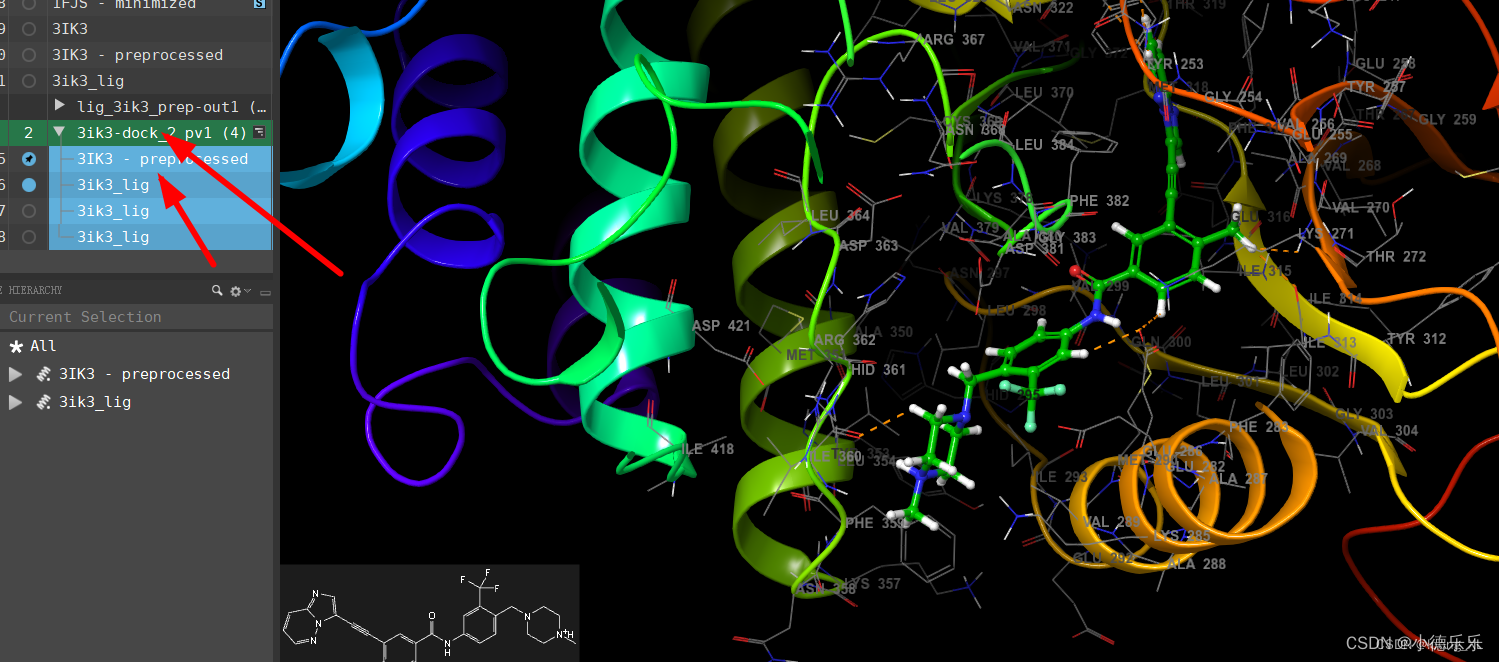
4、生成grid结合口袋,模块Receptor Grid Generration,选择Molecule,然后点选下展示窗口的小分子
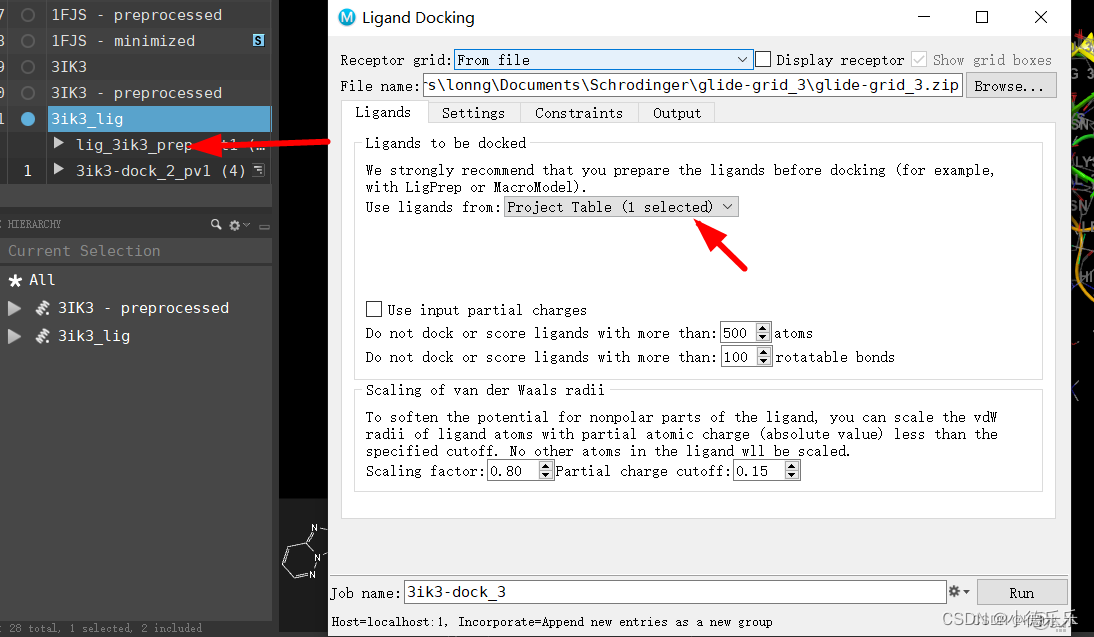
5、最后配体根据上面口袋对接,模块Ligand Docking,选择加载口袋zip文件,然后选择需要对接的优化过的配体,进行对接,结果会生成展示
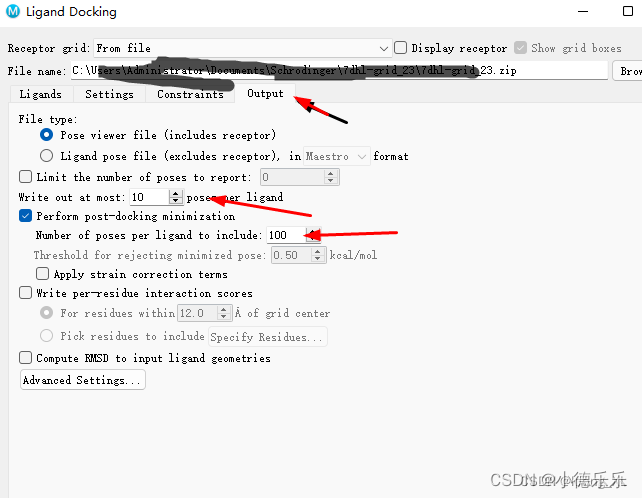
output这里10是表示输出对接数量,100是每个配体对接能量优化循环次数
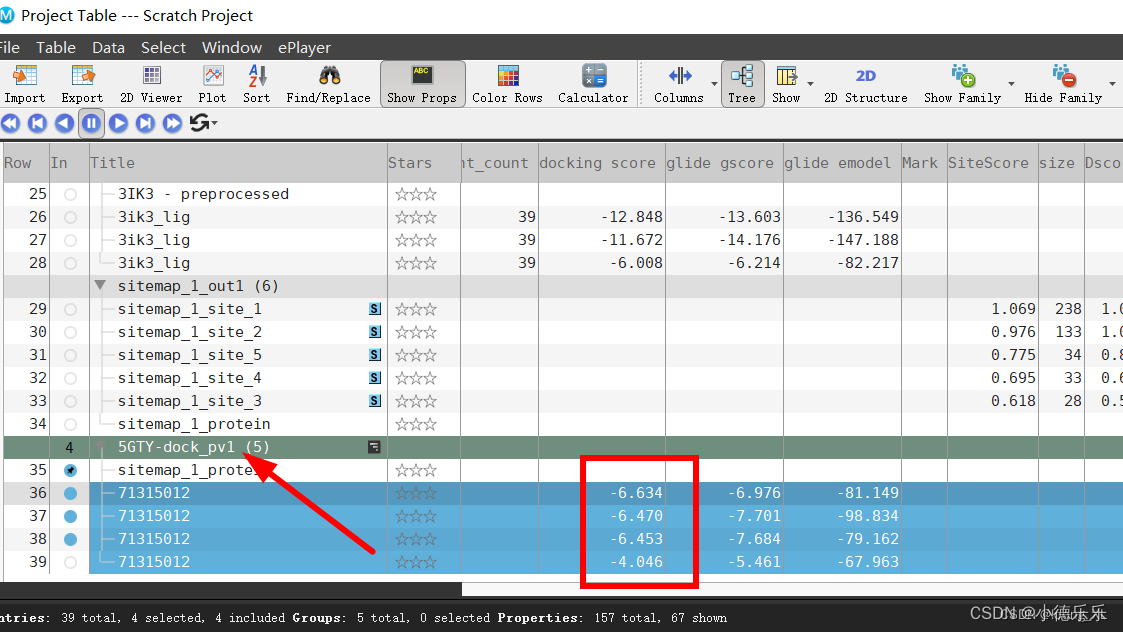
可以查看对接分数和结果:

二、对接蛋白受体无小分子配体情况
-
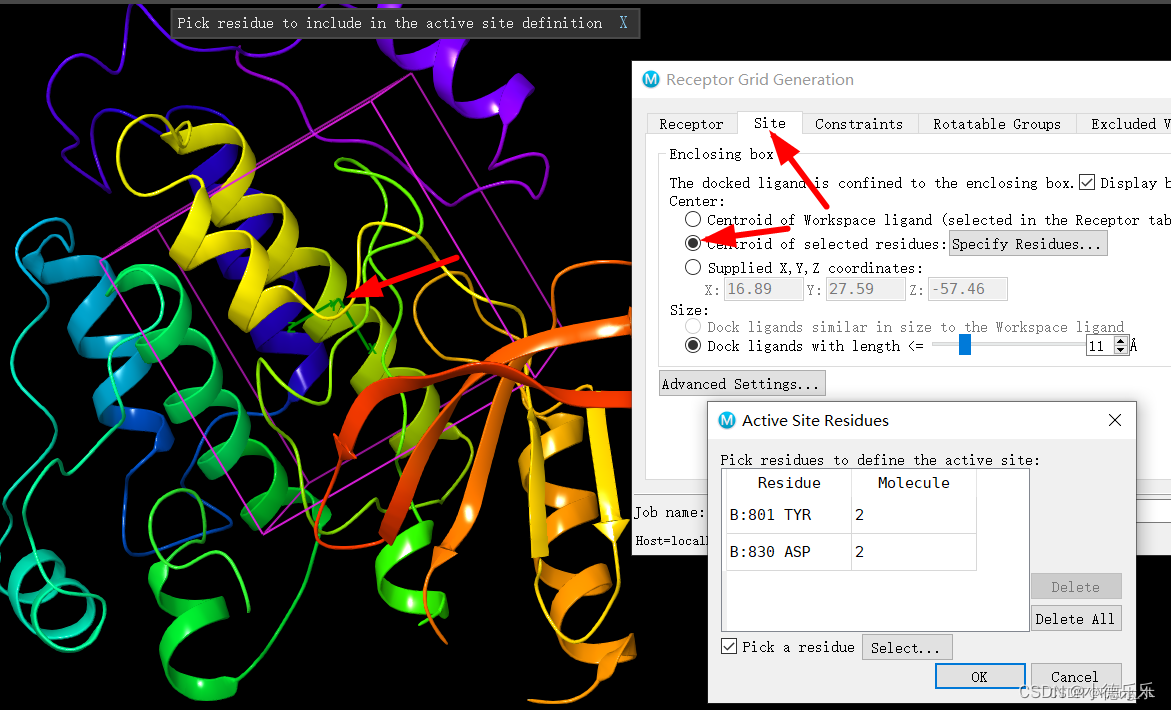
如果蛋白口袋已知,但没有小分子复合物,那可以通过模块Receptor Grid
Generration里选择对应蛋白口袋附件的氨基酸残基来生产口袋,再Site下选择第二个selected
residues,然后鼠标点击展示区氨基酸残基确认口袋选择;然后其他步骤都一样没有变化;
-
通过SiteMap模块自动查询可能的口袋进行对接;
1)一个不同与上面不同主要是这里的对接口袋需要通过其他方法去生成,这里用的模块是SiteMap,他会计算出蛋白受体可能的对接口袋;
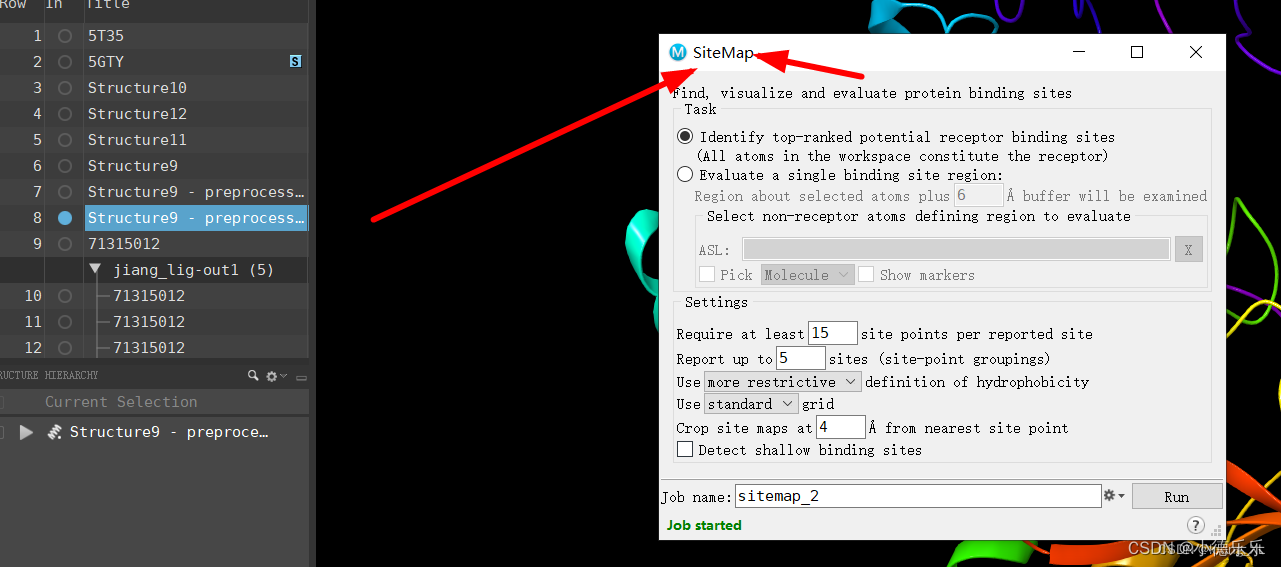
生成了5个可能口袋,按ctrl可以多选展示

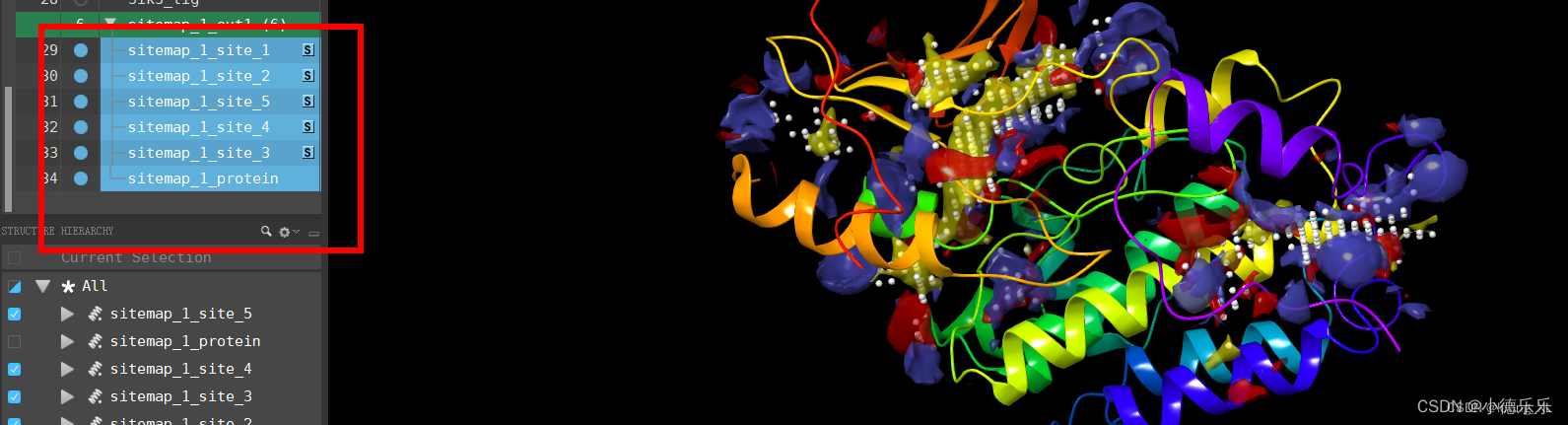
2)第二个不同是Grid生成,模块还是Receptor Grid Generration,但这里选择Entry,然后点选下展示上面一步产生的可能口袋位置;
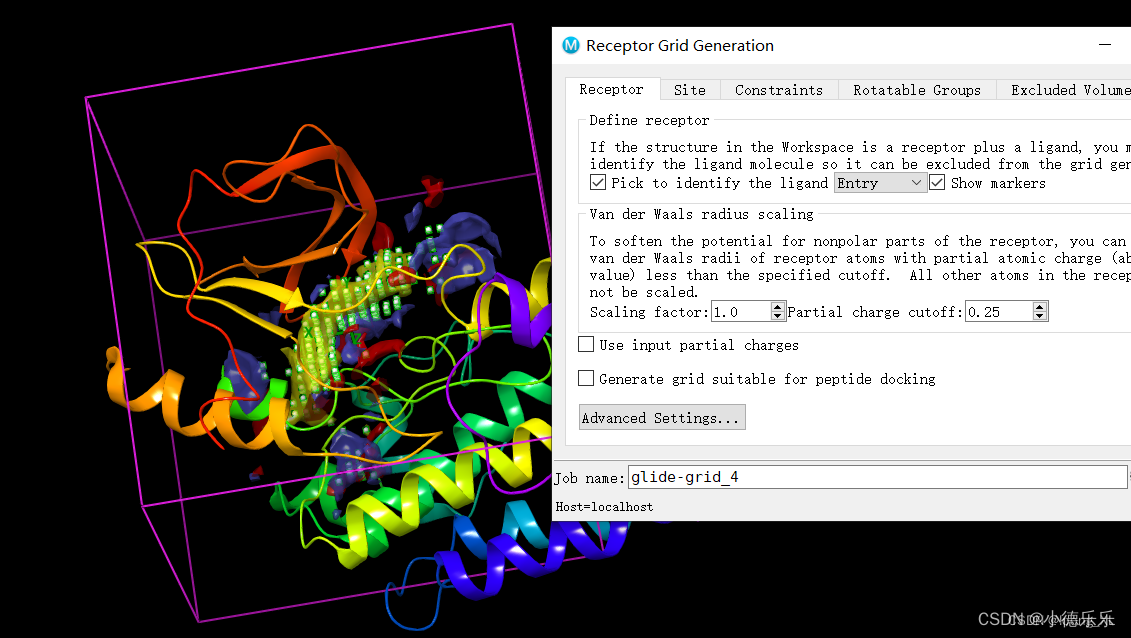
这里可以进行选择框大小自定义
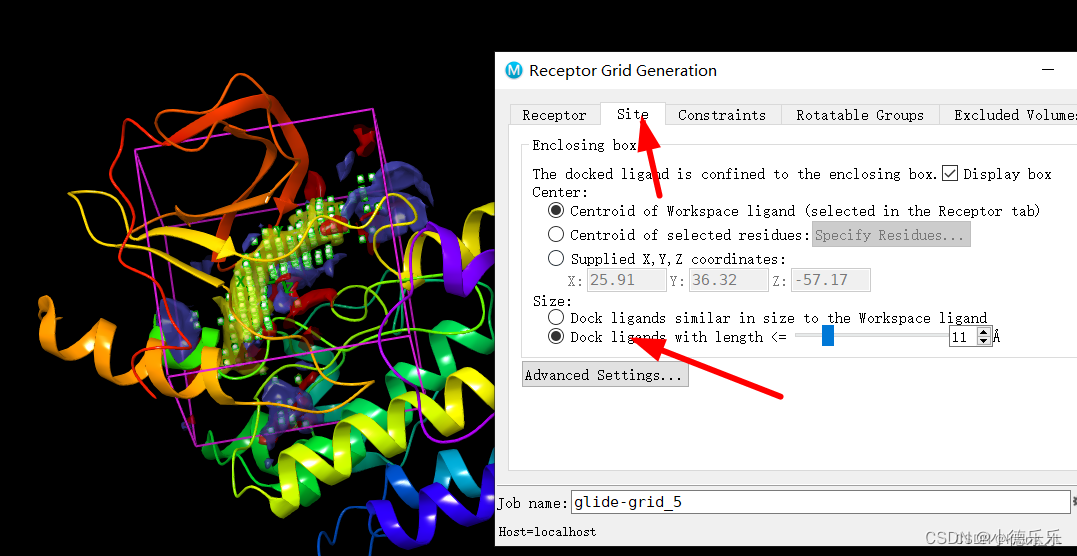
3)除上面两部区别,其他步骤,小分子与蛋白准备,对接步骤都是一样的
最后对接结果展示:
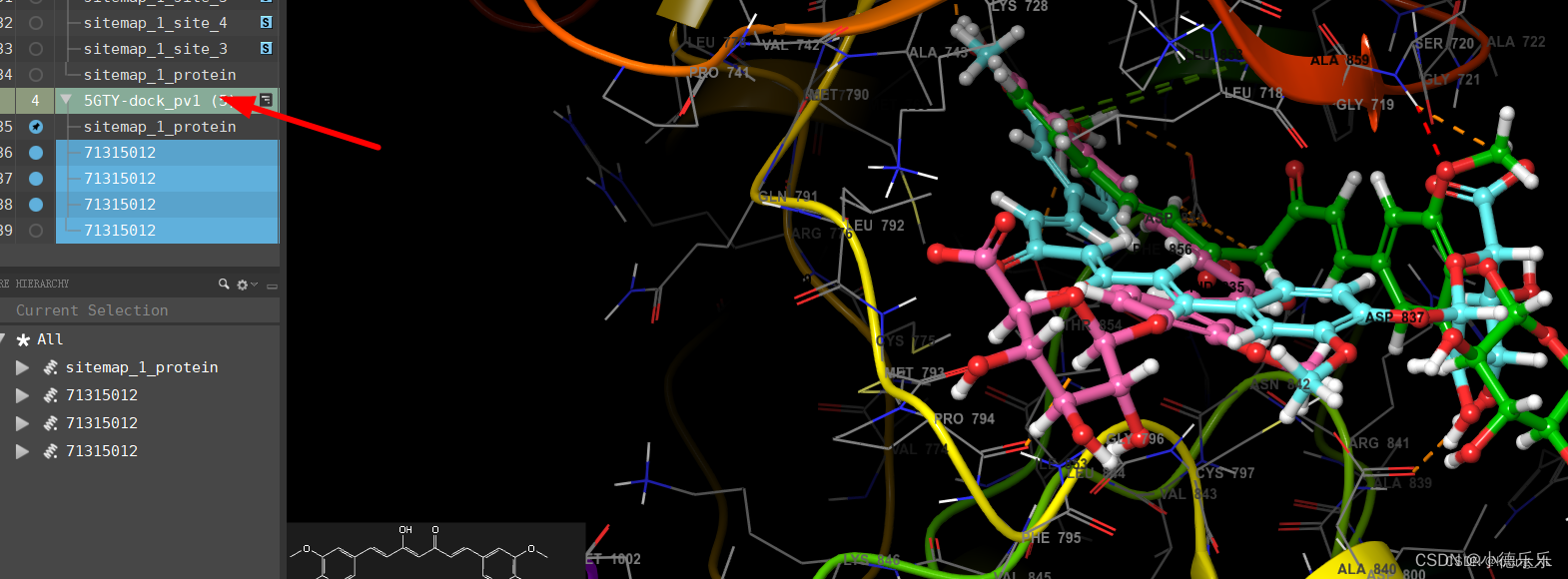
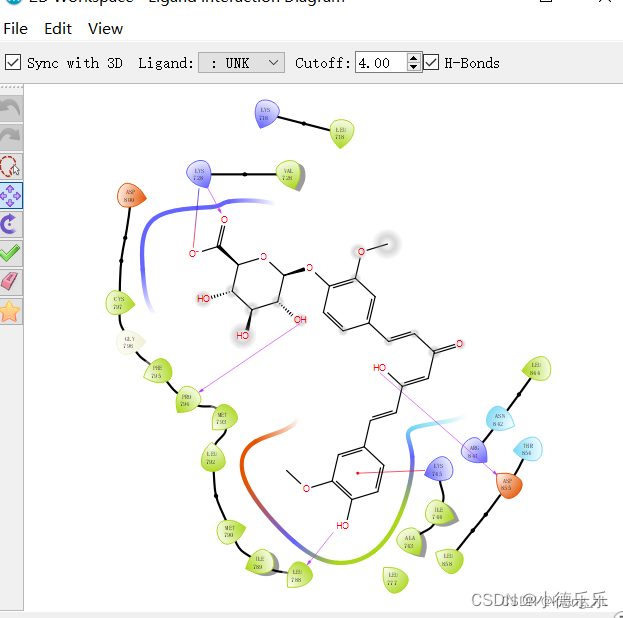
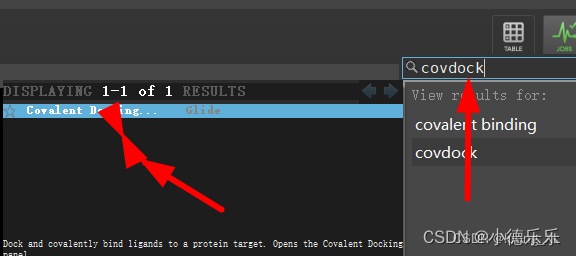
3、共价对接Covalent Docking 模块
参考:
- https://www.bilibili.com/video/BV1xL4y1n7Bn/
- https://blog.csdn.net/monkeny/article/details/127437406
.
- 博主:loong_XL——https://blog.csdn.net/weixin_42357472/article/details/128693565
更多推荐
 已为社区贡献1条内容
已为社区贡献1条内容









所有评论(0)